当我们讨论k-means算法时, 我们看到我们必须给出簇数作为输入参数之一. 在现实世界中, 我们不会提供这些信息. 我们绝对可以扫描参数空间, 使用轮廓系数得分找出最佳聚类数, 但这将是一个昂贵的过程! 如果有一种方法可以告诉我们数据中的集群数量, 那不是很好吗? 这就是噪声应用(DBSCAN)的基于密度的空间聚类.
这通过将数据点视为密集群组来实现. 如果一个点属于一个集群, 则应该有很多属于同一个集群的其他点, 我们可以控制的一个参数是这个点与其他点的最大距离 这被称为epsilon. 特定集群中的两点不应超过epsilon. 您可以在http://staffwww.itn.liu.se/~aidvi/courses/06/dm/Seminars2011/DBSCAN(4).pdf上了解更多信息. 这种方法的主要优点之一是它可以处理异常值. 如果在低密度区域中有一些点, DBSCAN将会将这些点作为异常值来检测,而不是将其强制为集群.
怎么做...?
- 这个食谱的完整代码在estimate_clusters.py文件. 我们来看看它是如何构建的. 创建一个新的Python文件, 并导入必需的包:
from itertools import cycle
import numpy as np
from sklearn.cluster import DBSCAN
from sklearn import metrics
import matplotlib.pyplot as plt
from utilities import load_data
- 导入数据:
input_file = 'data_perf.txt'
X = load_data(input_file)
- 我们需要找到最好的参数. 我们来初始化一些变量:
# Find the best epsilon
eps_grid = np.linspace(0.3, 1.2, num=10)
silhouette_scores = []
eps_best = eps_grid[0]
silhouette_score_max = -1
model_best = None
labels_best = None
- 让我们扫描参数空间
for eps in eps_grid:
# Train DBSCAN clustering model
model = DBSCAN(eps=eps, min_samples=5).fit(X)
# Extract labels
labels = model.labels_
# Extract performance metric
silhouette_score = round(metrics.silhouette_score(X, labels), 4)
silhouette_scores.append(silhouette_score)
print ("Epsilon:", eps, " --> silhouette score:", silhouette_score)
if silhouette_score > silhouette_score_max:
silhouette_score_max = silhouette_score
eps_best = eps
model_best = model
labels_best = labels
- 我们来绘制柱状图
# Plot silhouette scores vs epsilon
plt.figure()
plt.bar(eps_grid, silhouette_scores, width=0.05, color='k', align='center')
plt.title('Silhouette score vs epsilon')
# Best params
print ("Best epsilon =", eps_best)
- 让我们存储最好的模型和标签
# Associated model and labels for best epsilon
model = model_best
labels = labels_best
- 某些数据点可能仍未分配. 我们需要识别它们, 如下所示:
# Check for unassigned datapoints in the labels
offset = 0
if -1 in labels:
offset = 1
- 提取集群的数量
# Number of clusters in the data
num_clusters = len(set(labels)) - offset
print ("Estimated number of clusters =", num_clusters)
- 我们需要提取所有核心样品:
# Extracts the core samples from the trained model
mask_core = np.zeros(labels.shape, dtype=np.bool)
mask_core[model.core_sample_indices_] = True
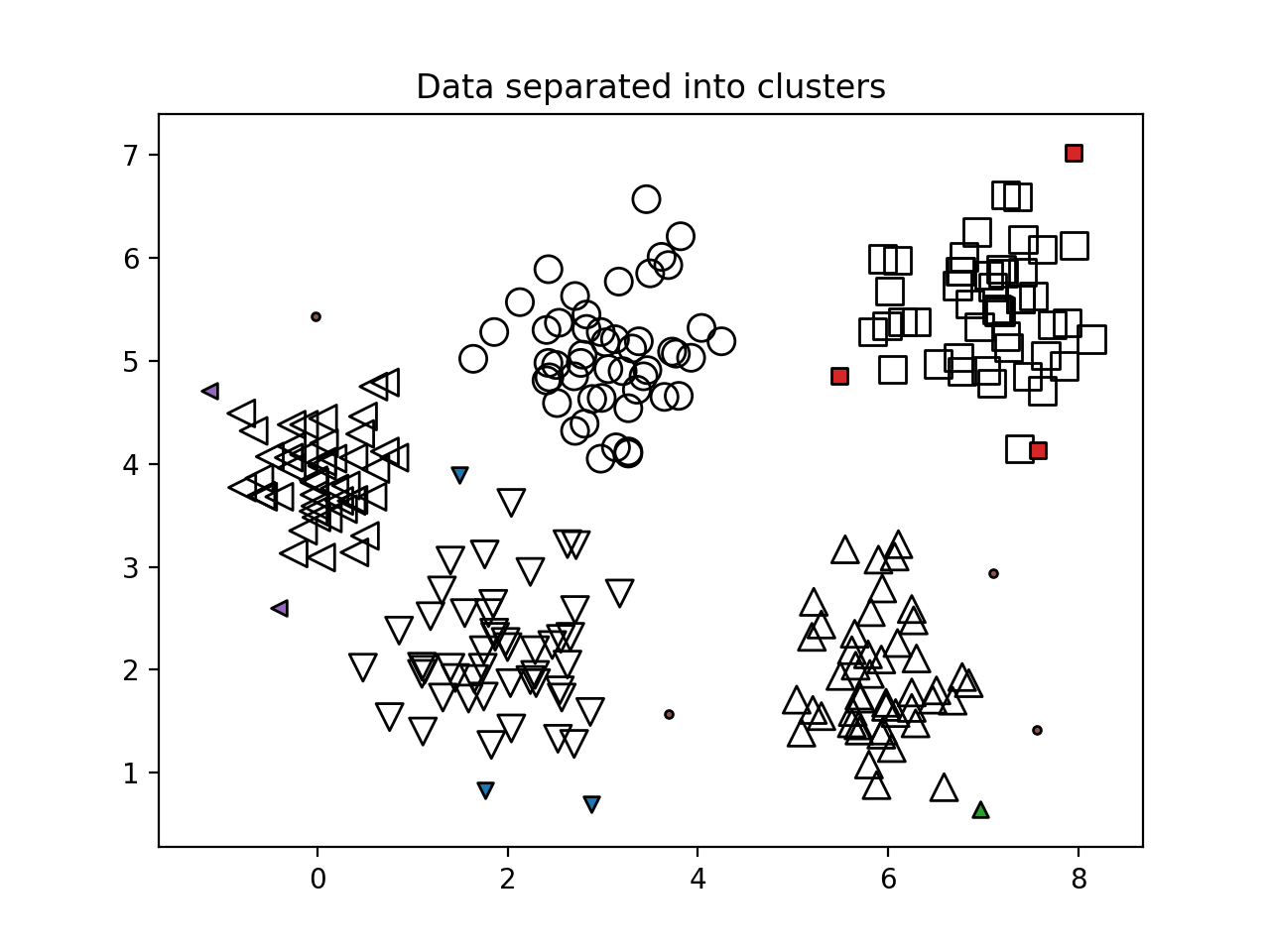
- 我们来看看结果集. 我们将首先提取一组唯一标签并指定不同的标记:
# Plot resultant clusters
plt.figure()
labels_uniq = set(labels)
markers = cycle('vo^s<>')
- 我们迭代集群, 并使用不同的标记绘制数据点:
for cur_label, marker in zip(labels_uniq, markers):
# Use black dots for unassigned datapoints
if cur_label == -1:
marker = '.'
# Create mask for the current label
cur_mask = (labels == cur_label)
cur_data = X[cur_mask & mask_core]
plt.scatter(
cur_data[:, 0],
cur_data[:, 1],
marker=marker,
edgecolors='black',
s=96,
facecolors='none'
)
cur_data = X[cur_mask & ~mask_core]
plt.scatter(
cur_data[:, 0],
cur_data[:, 1],
marker=marker,
edgecolors='black',
s=32
)
plt.title('Data separated into clusters')
plt.show()
- 运行代码会得到如下结果:
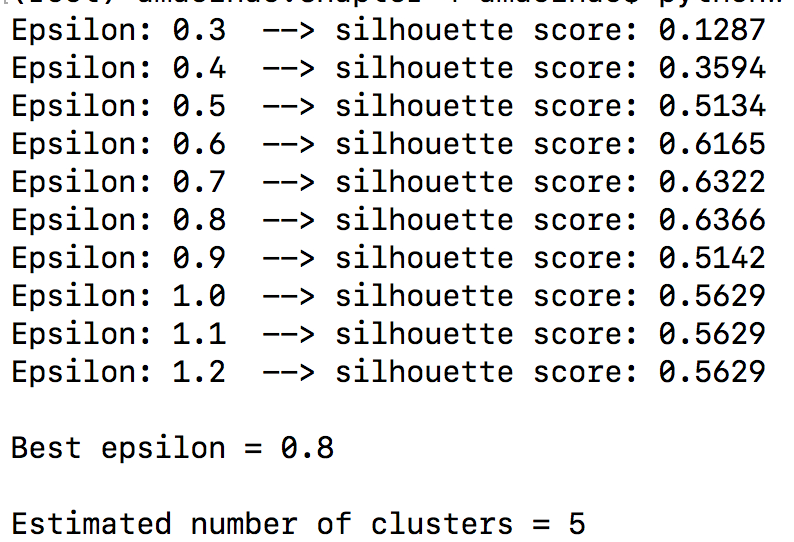
柱状图如下:
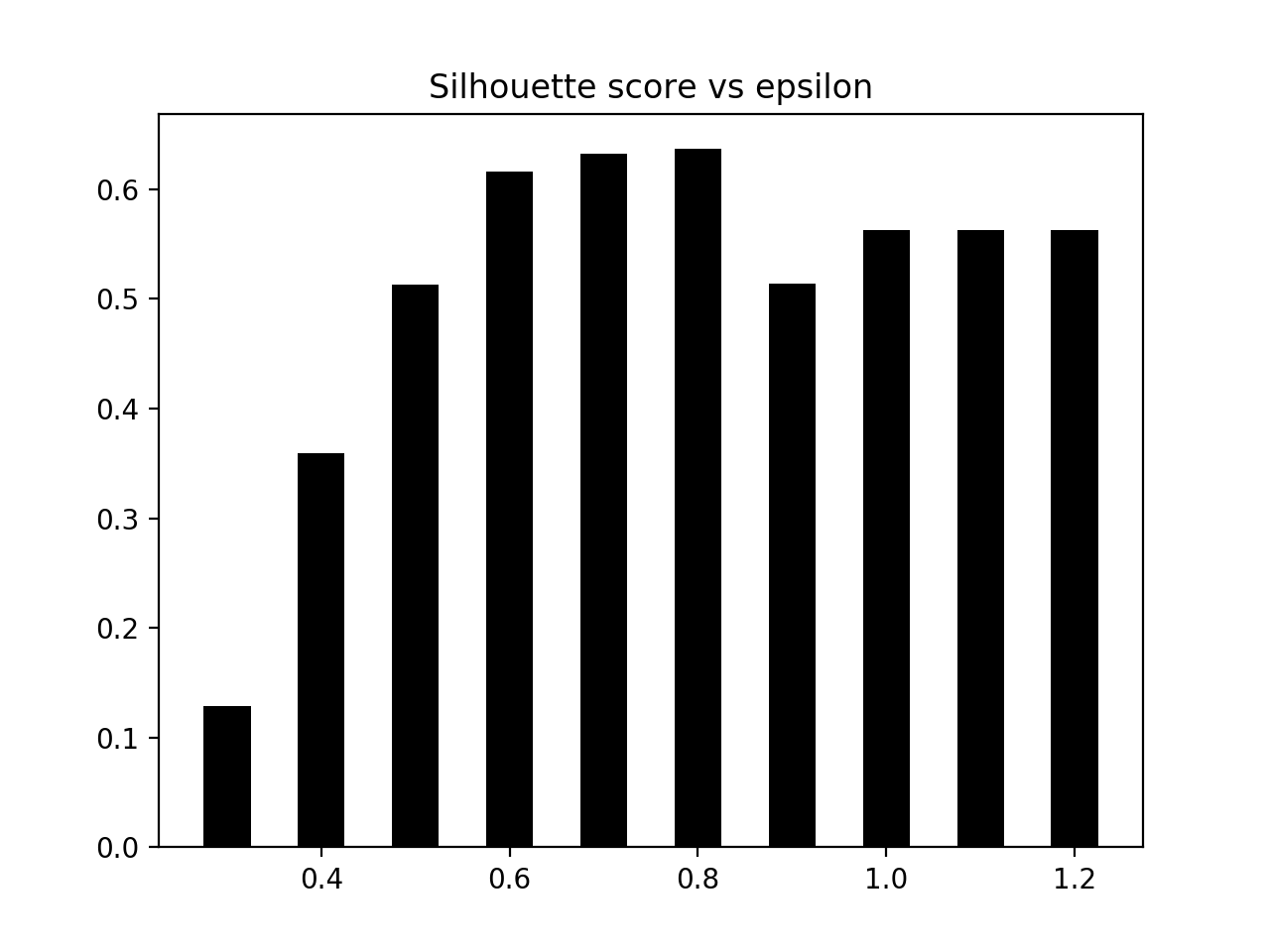
我们来看看标记的数据点以及未分配的数据点, 如下图所示: